환자유래모델 시 유전자 오류 줄여
인간-생쥐 유전자 섞이는 돌연변이 검출
[그린포스트코리아 이재형 기자] 국내 연구진이 유전자 분석을 통해 암 진료 시 정확도를 높이는 기술을 고안했다.
김상우 교수(연세대 의대) 연구팀이 환자의 암세포를 분석할 때 외부요인을 줄여 분석의 정확도를 높이는 방법을 개발했다고 과학기술정보통신부가 11일 밝혔다.
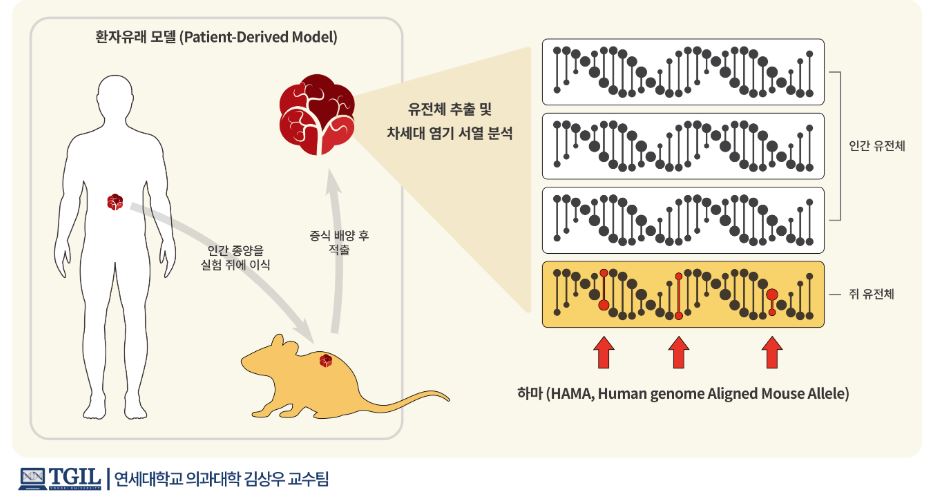
병원에서 암을 치료할 때 유전자검사나 약물반응검사 등을 위해 암 종양조직을 거듭 분석해야 하나 매번 환자 몸에서 조직을 뜯어낼 순 없었다. 이에 의료계에선 채취 한 종양세포를 증식시켜 여러 검사의 시료로 쓸 수 있게 하는 환자유래모델(PDMS, patient-derived models)을 사용해왔다.
그러나 환자유래모델은 종양세포를 보존할 때 생쥐의 체내에 이식해 증식시키거나, 생쥐의 세포와 함께 배양하는 탓에 오류가 났다. 분석 과정에서 환자의 세포와 쥐의 세포가 섞여 함께 자라다보니 적게는 10%, 많게는 70%까지 함께 자라기 때문이다.
이에 연구진은 환자유래모델에서 배양한 세포를 분석해 돌연변이 분석 오류를 파악하고 방지하는 방법을 개발했다.
연구진은 쥐와 사람의 유전체와 섞여서 유전자 변이가 발생할 때 나타나는 120만가지 DNA 현상을 ‘하마’(HAMA, human-genome aligned mouse allele)라고 명명하고, ‘하마’를 통해 생쥐의 유전체 정보로 인한 오류가능성을 확인할 수 있게 했다.
또 유전체 검사 데이터를 통해 나오는 ‘하마’의 비율을 분석해 환자유래모델에 쥐 세포가 얼마나 섞여있는지 비율까지 계산할 수 있는 방법을 제시했다.
또 150가지가 넘는 가상의 오염 데이터를 비교 분석해 최적의 오염 배제 방법을 알아냈다. 이 방식을 최적 유전자분석법에 적용한 결과 기존 분석 대비 정확성을 약 58% 가량 높일 수 있었다고 연구진은 설명했다.
김상우 교수는 “본 연구는 체외에서 보존, 증식된 환자 암세포 시료(Specimen)의 유전체 분석과정에서 발생할 수 있는 오류를 바로잡아 향후 더욱 정확한 정보에 기초하여 환자를 치료할 수 있는 실마리가 될 것”이라고 밝혔다.
과학기술정보통신부 개인기초연구(중견연구) 사업의 지원으로 수행된 이번 연구의 성과는 유전체학 분야 국제학술지 ‘지놈 바이올로지(Genome Biology)’에 11월 11일 자로 게재되었다.
silentrock91@greenpost.kr